Control de los procesos biotecnológicos
Hay una gran cantidad de desafíos en el monitoreo y control de los procesos biotecnológicos donde el análisis exploratorio de datos, la agrupación y la clasificación no cumplen con los requisitos de la aplicación específica. Por ejemplo, en los sensores virtuales (ver Capítulo 15), se requieren métodos de regresión que permitan predecir un atributo de calidad crítico deseado (CQA) o un Parámetro de Proceso Crítico (CPP) a partir de datos del proceso. En un mundo ideal, estos datos se obtendrían mediante mediciones analíticas oportunas, precisas y robustas. Sin embargo, la falta de técnicas apropiadas para medir CQAs, especialmente cuando están presentes en cantidades muy bajas en medios complejos, plantea la necesidad de estimación inferencial a través de la tecnología de sensores virtuales (ver Capítulo 15) de tales atributos y CPPs utilizando mediciones del proceso que están fácilmente disponibles. Esta parte del capítulo, por lo tanto, revisa brevemente los métodos de regresión establecidos, destacando especialmente sus aplicaciones en la modelización de biorreactores.
La técnica de regresión lineal más ampliamente utilizada para correlacionar variables causales (X) y variables de salida (Y) en una variedad de procesos es el método de mínimos cuadrados parciales (PLS, por sus siglas en inglés) y sus diversas modificaciones. El algoritmo PLS se basa en la proyección de las variables de entrada (X) y salida (Y) en un número de variables latentes e identifica la correlación de mínimos cuadrados entre estas nuevas variables mediante una regresión lineal de entrada única y salida única.
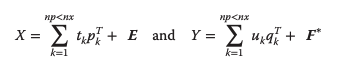
Donde E y F son matrices residuales, np es el número de componentes internos que se utilizan en el modelo, y nx es el número de variables causales.
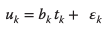
Donde bk es un coeficiente de regresión y 𝜀k se refiere al error de predicción. El desafío asociado con control de los procesos biotecnológicos y conjuntos de datos grandes recopilados utilizando métodos de medición multianalito (por ejemplo, métodos espectroscópicos u «ómicos») se aborda en parte gracias a la capacidad de PLS para reducir las dimensiones de los datos. Sin embargo, la evidencia muestra que la preselección de variables para el modelado de PLS puede mejorar significativamente el rendimiento del modelo. Por ejemplo, el PLS ortogonalizado (O-PLS) elimina la variación ortogonal con respecto a las variables de salida Y del conjunto de variables de entrada X. Utilizando este método, Yang et al. demostraron que la variabilidad óptica no relevante para los analitos en los espectros NIR de tejidos humanos les permitió tener en cuenta la superposición de tejidos y, por lo tanto, obtener una representación más precisa de las concentraciones de analitos dentro de un tejido específico.
La limitación reportada debido a la suposición de relaciones lineales entre las variables se aborda en una variedad de métodos alternativos de PLS. Estos incluyen la incorporación de relaciones polinómicas en la estructura de PLS, el uso de redes neuronales artificiales como modelos internos de PLS o estructuras híbridas que incorporan ecuaciones de balance de masas basadas en una comprensión de primeros principios del proceso. Al igual que con el control de los procesos biotecnológicos, los métodos de extracción de características y clasificación, las redes neuronales representan un enfoque alternativo de modelado de regresión no lineal con una amplia gama de ejemplos de aplicación en la modelización y monitoreo de procesos biotecnológicos.
Extracción de características utilizando PCA
Se presenta un ejemplo de la aplicación de la técnica de extracción de características en el control de los procesos biotecnológicos para identificar las diferencias en el rendimiento del biorreactor bajo diversas condiciones utilizando medidas de nariz electrónica para demostrar la utilidad de enfoques como este. Para este estudio de caso, se llevaron a cabo una serie de seis fermentaciones de E. coli recombinante y se utilizaron para investigar el efecto de los regímenes de inducción en la producción de 𝛽-galactosidasa. Durante estas cultivaciones, se utilizó una nariz electrónica híbrida comercial de la antigua Nordic Sensor Technologies A.B, Linköping (posteriormente Applied Sensors), NST 3320. La formación de 𝛽-galactosidasa en todos los lotes se indujo con isopropil-𝛽-d-tiogalactopiranósido (IPTG).
Dos de los lotes, denominados 1A y 1B, fueron inducidos temprano (durante la fase de crecimiento exponencial a las 19 horas de registro). Otros dos lotes, referidos como 2A y 2B, fueron inducidos durante la fase estacionaria a las 26-27 horas de registro. La cepa de E. coli con el vector de expresión operado por el promotor lac UV se utilizó en los cuatro de estos lotes. Las dos cultivaciones restantes, 3A y 3B, se llevaron a cabo como control de los procesos biotecnológicos. El lote 3A se indujo temprano a las 20 horas de registro y el lote 3B se indujo tarde a las 27 horas de registro. En estos dos lotes se utilizó una cepa que portaba un plásmido vacío, lo que resultó en la ausencia de producción de 𝛽-galactosidasa. Se midieron datos extensos en línea y fuera de línea, incluyendo temperatura, presión, pH, tasa de flujo de gas, porcentaje de O2 y CO2 en el gas de escape y porcentaje de oxígeno disuelto en el caldo, concentraciones de glucosa, ácido acético y formato, densidad óptica (600 nm) y peso en seco.
Cinco de los sensores de la nariz electrónica, concretamente aquellos sensibles al hidrógeno, metano y humedad, fueron eliminados del conjunto de datos antes del análisis debido a su falta de relevancia en esta aplicación.
Se realizó un análisis de componentes principales (PCA) en la parte de Respuesta de las señales de sensor restantes y se muestran los gráficos de puntuaciones para los datos de Respuesta sin procesar de los seis lotes. Esto se compara con el mismo análisis después de eliminar las variables insignificantes (lo que reduce la dimensionalidad de los datos de 120 variables a 49 variables).
En estas figuras, las puntuaciones de cada uno de los lotes se muestran en diferentes colores utilizando símbolos diferentes (ver leyenda). Claramente, la reducción de variables no ha afectado la capacidad del PCA para discriminar entre los lotes individuales. Los lotes que destacan por tener un perfil de puntuaciones diferente, es decir, 1A, 2A y en cierta medida 1B, fueron, por lo tanto, analizados en una comparación par a par para elucidar las diferencias observadas.

